Các nhà sinh học tổng hợp ngày càng trở nên sáng tạo trong kỹ thuật chế tạo men hoặc vi khuẩn để tạo ra các hóa chất hữu ích—từ nhiên liệu đến vải và thuốc—ngoài khả năng thông thường của vi khuẩn.
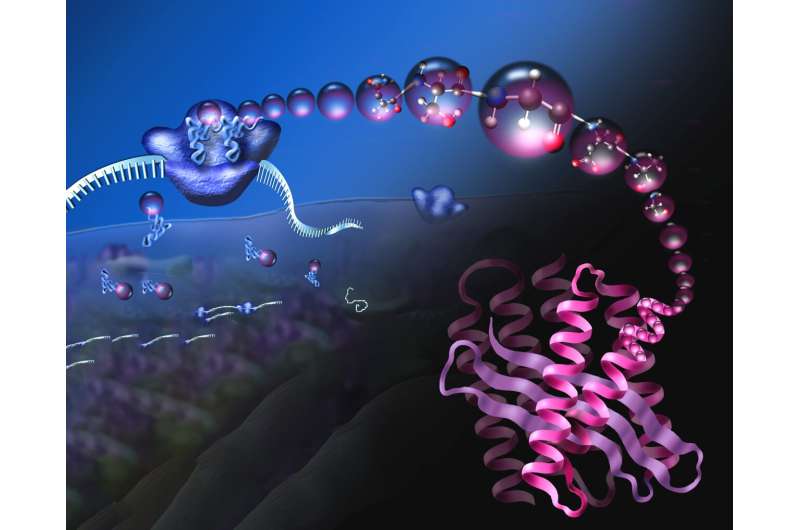
Nhưng một nhóm các nhà hóa học ở nhiều trường đại học có một mục tiêu đầy tham vọng hơn: trang bị lại các nhà máy sản xuất polypeptide của tế bào—các ribosome quay các axit amin thành protein—để tạo ra các chuỗi polymer phức tạp hơn những gì hiện có thể tạo ra trong một tế bào hoặc một tế bào. ống nghiệm.
Doanh nghiệp nghiên cứu có trung tâm tại Đại học California, Berkeley, hiện đang báo cáo tiến độ đáng kể hướng tới mục tiêu đó, bằng chứng là ba bài báo mới giải quyết ba rào cản lớn: cách lập trình lại các tế bào để cung cấp cho ribosome các khối xây dựng khác ngoài alpha- amino . axit tạo nên tất cả các loại protein ngày nay; làm thế nào để dự đoán khối xây dựng nào tạo ra chất nền tốt nhất; và cách điều chỉnh ribosome để kết hợp các khối xây dựng mới này thành polyme.
Mục tiêu cuối cùng của Trung tâm Khoa học Quốc gia về Vật liệu Mã hóa Di truyền (C-GEM) là làm cho hệ thống dịch mã có thể lập trình đầy đủ, để đưa các hướng dẫn mRNA vào tế bào cùng với các khối xây dựng mới—chứ không phải axit alpha-amino được tìm thấy ngày nay— sẽ cho phép ribosome tạo ra vô số chuỗi phân tử mới. Những chuỗi này có thể tạo cơ sở cho vật liệu sinh học mới, enzyme mới, thậm chí cả thuốc mới.
Các bài báo, xuất hiện trên các tạp chí Nature Chemistry và ACS Central Science , là phần mở đầu của một cuốn sách hướng dẫn tái cấu trúc bộ máy tổng hợp tế bào để tạo ra các polyme chưa từng thấy, bao gồm polyme sinh học và polyme vòng, được gọi là chu trình lớn peptit, với các ứng dụng được xác định trước hoặc hoàn toàn không lường trước được.
"C-GEM đang nghiên cứu để sinh tổng hợp các phân tử chưa từng được tạo ra trong tế bào trước đây và được thiết kế để có các đặc tính độc đáo. Các công cụ này có thể được các nhà hóa học polyme, hóa dược và nhà khoa học vật liệu sinh học áp dụng rộng rãi để tạo ra các vật liệu riêng biệt với các chức năng mới, " Giám đốc C-GEM Alanna Schepartz, Chủ tịch xuất sắc của TZ và Irmgard Chu về Hóa học và là giáo sư sinh học phân tử và tế bào tại UC Berkeley cho biết. "Mục tiêu cuối cùng là mở rộng chức năng và tính linh hoạt của protein và polypeptide, cả về vật liệu và dược phẩm."
Cô ấy nói, một ví dụ sẽ là lập trình cho ribosome tổng hợp một loại polymer là vật liệu lai giữa tơ nhện - một trong những protein tự nhiên cứng nhất - và nylon, một loại polymer hiện được sản xuất trong các buồng phản ứng hóa học. Mặc dù tơ nhện hiện có thể được tạo ra từ các vi khuẩn biến đổi gen, nhưng công nghệ mà C-GEM đang phát triển có thể cho phép các vi khuẩn tương tự tạo ra vô số polyme pha trộn các khối xây dựng của tơ và nylon, tất cả chúng đều mới đối với các nhà hóa học và có các đặc tính độc đáo. Công nghệ này cũng có thể được sử dụng để tạo ra các polyme giống như protein có khả năng chịu nhiệt tốt hơn so với các protein tự nhiên.
Một khía cạnh mạnh mẽ của cỗ máy ribosome có thể lập trình có thể tổng hợp polyme là nó cho phép các nhà nghiên cứu phát triển polyme để hoàn thiện hoạt động của chúng, giống như protein đã tiến hóa qua hàng trăm triệu năm để cải thiện khả năng hoạt động của tế bào và sinh vật.
Jamie Cate, giáo sư hóa học của UC Berkeley cho biết: “Chúng tôi đã có các polyme protein phát triển trên hành tinh trong hàng tỷ năm, nhưng chúng tôi đã bị hạn chế về những polyme đó là gì vì các khối xây dựng là 20 axit amin giống nhau”. sinh học phân tử và tế bào. "Nếu chúng ta có thể phát triển một hệ thống mà bạn thực sự có thể áp dụng quá trình tiến hóa cho những polyme mới này, thì nó giống như một nền tảng mà bất kỳ ai có ý tưởng sáng tạo đều có thể sử dụng để phát triển polyme thành thứ họ muốn."
Một hệ thống như vậy được xây dựng dựa trên sự phát triển có định hướng của các enzyme protein mà Frances Arnold, cựu sinh viên UC Berkeley, đã nhận được giải Nobel Hóa học năm 2018.
“Đó là một bước tiến xa hơn những gì Frances Arnold đã làm trong việc phát triển quá trình tiến hóa có định hướng,” Cate nói. "Cô ấy đã phát triển quá trình tiến hóa có định hướng cho protein. Những gì chúng tôi đang cố gắng thực hiện là thiết lập một cách mà bạn có thể làm điều này đối với các polyme chưa từng tiến hóa trong tự nhiên trước đây."
Kỹ thuật một ribosome hoàn toàn mới
Trong tất cả các tế bào, protein được lắp ráp bởi một cỗ máy nano, ribosome, tiếp nhận các chỉ dẫn từ một phân tử ARN được gọi là ARN thông tin (mRNA)—mARN giống như một bản sao hoạt động của mã DNA của gen—và đọc các chỉ dẫn đó để lắp ráp một protein, axit amin bằng axit amin. Thật đáng ngạc nhiên, chuỗi protein tuyến tính hầu như luôn gấp lại thành một cấu trúc 3D được xác định rõ ràng, sẵn sàng phục vụ mục đích tiến hóa của nó: như một enzym xúc tác cho các phản ứng trong tế bào, như một thành phần cấu trúc của tế bào hoặc như một chất điều chỉnh các hoạt động khác của tế bào .
Mười năm trước, việc trang bị lại cỗ máy nano phức tạp này dường như là điều không thể. Nhưng sự kiên trì của Schepartz trong việc thúc đẩy một dự án hoàn thành mục tiêu này đã dẫn đến C-GEM, tức là ba năm sau chu kỳ cấp vốn năm năm đầu tiên của nó.
Một trong những mục tiêu của trung tâm là cung cấp cho ribosome các khối xây dựng—cái gọi là monome—ngoài axit alpha-amino. Để đạt được mục tiêu này, nhóm C-GEM đã tập trung vào các enzym tải các đơn phân axit amin lên ARN vận chuyển (tRNA), các phân tử vận chuyển axit amin đến ribosome. Mỗi tRNA được mã hóa bằng vạch để cho biết nó mang loại axit nào trong số 20 axit amin.
Như đã báo cáo trong một bài báo Hóa học Tự nhiên được xuất bản vào ngày 1 tháng 6 và được đồng tác giả bởi Schepartz và các sinh viên tốt nghiệp Riley Fricke và Cameron Swenson, nhóm đã phát hiện ra rằng một họ tRNA synthetase có thể tải tRNA bằng bốn axit không phải alpha-amino khác nhau. Một trong số đó là khối xây dựng của nhiều phương pháp trị liệu polyketide khác nhau, bao gồm kháng sinh erythromycin và tetracycline.
Schepartz cho biết: “Chúng tôi đã xác định được các enzym tải tRNA bằng các đơn phân có cấu trúc khác với bất kỳ thứ gì đã được tải trên tRNA trước đây. "Một trong những monome là tiền chất có thể được sử dụng để lắp ráp các phân tử giống như polyketide. Các nhà khoa học đã cố gắng trong nhiều thập kỷ để tái cấu trúc các mô-đun tổng hợp polyketide nhằm tạo ra các thư viện sản phẩm tự nhiên. Những nghiên cứu này đã dạy cho chúng tôi rất nhiều điều về sự phức tạp của các mô-đun này , nhưng phần kỹ thuật đã rất khó."
Các monome mới được ribosome bản địa ở vi khuẩn E. coli sẵn sàng chấp nhận , chứng minh rằng có thể kết hợp các loại hóa chất khác nhau vào polyme protein toàn axit amin thông thường.
"Kháng kháng sinh là một vấn đề lớn," cô nói thêm. "Nếu chúng ta có thể giúp giải quyết vấn đề đó bằng cách tạo ra các phân tử mới có chức năng mã hóa các phương thức hoạt động độc đáo, thì đó sẽ là một đóng góp to lớn."
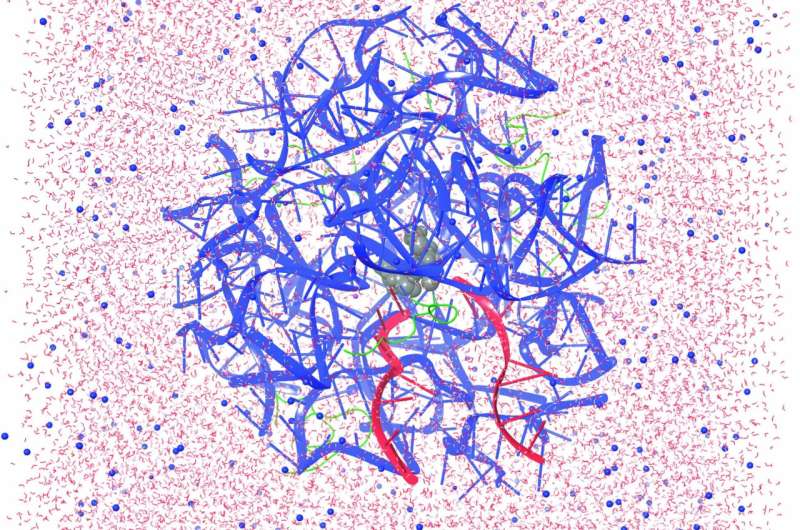
Trong một bài báo thứ hai, xuất hiện vào ngày 30 tháng 5 trên ACS Central Science , tác giả chính và nghiên cứu sinh sau tiến sĩ Chandrima Mujumdar, cùng với Cate và Schepartz, đã sử dụng kính hiển vi điện tử đông lạnh (cryo-EM) để thu được cấu trúc chi tiết của ba đơn phân có liên quan—không có đơn phân tử nào trong số đó là alpha. -axit amin—liên kết với ribosome của E. coli. Các chi tiết cho thấy cách các đơn phân này liên kết—mặc dù kém hơn nhiều so với các axit amin—và cung cấp các gợi ý về cách thay đổi các đơn phân hoặc ribosome để cải thiện khả năng của ribosome trong việc sử dụng chúng để tạo ra các polyme mới.
Trong một bài báo thứ ba, xuất hiện vào ngày 12 tháng 6 trên tạp chí Nature Chemistry , Cate, Schepartz và tác giả chính Zoe Watson, một nghiên cứu sinh sau tiến sĩ, báo cáo cấu trúc cryo-EM của ribosome E. coli trong khi liên kết các axit alpha-amino bình thường. Đối với bài báo này, nhóm đã hợp tác với công ty Schrödinger Inc. ở San Diego, công ty sử dụng máy tính để lập mô hình liên kết protein. Ara Abramyan của Schrodinger đã sử dụng cấu trúc cryo-EM làm điểm khởi đầu để chạy các mô phỏng siêu động lực học nhằm giúp hiểu được monome phi tự nhiên nào sẽ phản ứng trong trung tâm xúc tác của ribosome —trung tâm peptidyl transferase (PTC)—và cái nào sẽ không phản ứng.
Schepartz và Cate nhấn mạnh rằng tất cả những điều chỉnh này đối với hệ thống ribosome phải hoạt động bên trong một tế bào sống độc lập với các ribosome bình thường để việc sản xuất các polyme mới không cản trở quá trình sản xuất protein hàng ngày cần thiết cho sự sống.
Schepartz nói: “Chúng tôi muốn các enzyme – synthetase – và ribosome có thể được sử dụng trong tế bào, bởi vì đó là cách hoạt động này sẽ có thể mở rộng. "Mục tiêu đó đòi hỏi các ribosome mạnh mẽ, các enzym tuyệt vời và nhiều hiểu biết về hóa học về cách thức hoạt động của các cỗ máy phân tử phức tạp này. Đó là một vấn đề khó nhưng rất thú vị. Và chúng tôi có thể cho sinh viên và các nghiên cứu sinh sau tiến sĩ tiếp cận với một số ngành khoa học thực sự tuyệt vời."






